CRISPR遺伝子ノックアウト細胞株
VectorBuilderは、目的遺伝子(GOI)を永久的にノックアウトした安定細胞株を作製いたします。安定にノックアウトするために、CRISPR遺伝子編集技術を用います。GOIを標的とするシングルgRNAまたは一対のgRNAペアをCas9と共に細胞へ導入し、二本鎖切断(DSB)を生じさせます。これらの切断の修復は通常、フレームシフト変異や配列欠失を引き起こし、標的遺伝子の機能破壊をもたらします。
特長
- CRISPRの高い専門性: in vitroおよびin vivoにおける遺伝子編集技術ついて10年以上の経験を持つ弊社の専門チームが遺伝子ノックアウトを成功させる最適な遺伝子ターゲティング方法をデザインし、ご提案します。
- 非ウイルス性のデリバリー: エレクトロポレーションに基づくRNPデリバリーは効率的で安全、かつオフターゲット効果を最小限にします。
- 短い作業日数: ベクター設計から最短9週間でノックアウト細胞を樹立いたします。
サービス詳細
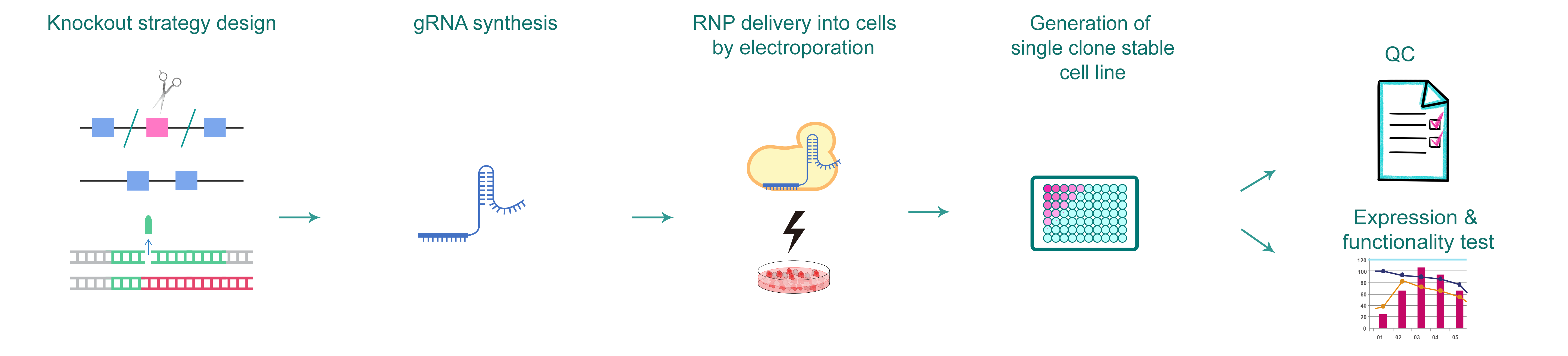
図1. CRISPRによる遺伝子ノックアウト細胞作製のワークフロー。
価格と作業日数 プライスマッチ
| サービス内容 | 納品形態 | 価格 (税別、送料別)* | 作業日数 |
|---|---|---|---|
| ノックアウト(フレームシフト変異) | ホモ接合体2クローン (>106 細胞/バイアル, 2本/クローン) | 620,000円より | 6-12 週間 |
| ノックアウト(配列欠損) | ホモ接合体 1クローン (>106 細胞/バイアル, 2本) | 930,000円より | 6-12 週間 |
* 追加のクローンまたはバイアルには追加料金がかかります。
QCアッセイ
| アッセイ内容 | 検査方法 |
|---|---|
| ノックアウト確認 (デフォルト) | PCR, サンガーシークエンス |
| 発現テスト (オプション) | RT-qPCR, WB, IF, FACS |
| オフターゲット検査 (オプション) | NGS, PCR, サンガーシークエンス |
| 染色体検査 (オプション) | 核型検査 |
| 無菌試験 (デフォルト) | マイコプラズマ検出(PCR), バイオバーデン試験 |
解析の追加
ご要望に応じて、増殖、アポトーシス、遊走、生存率、細胞毒性などのアッセイを含む、樹立細胞株の様々な表現型評価や機能検証を実施いたします。
ケーススタディ
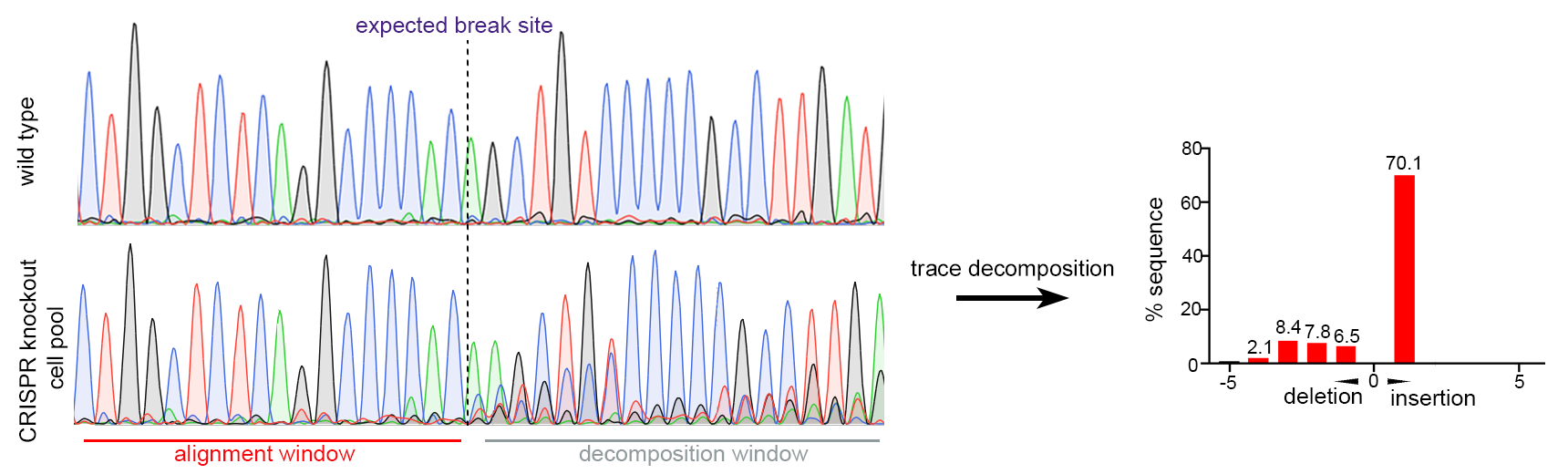
図2. Huh-7細胞におけるシングルgRNA CRISPR遺伝子ノックアウト。プールされた細胞のサンガーシークエンスにより、CRISPR標的部位周辺で効率的な変異導入が示され。trace decompositionにより97.6%の配列に変異があると推定。
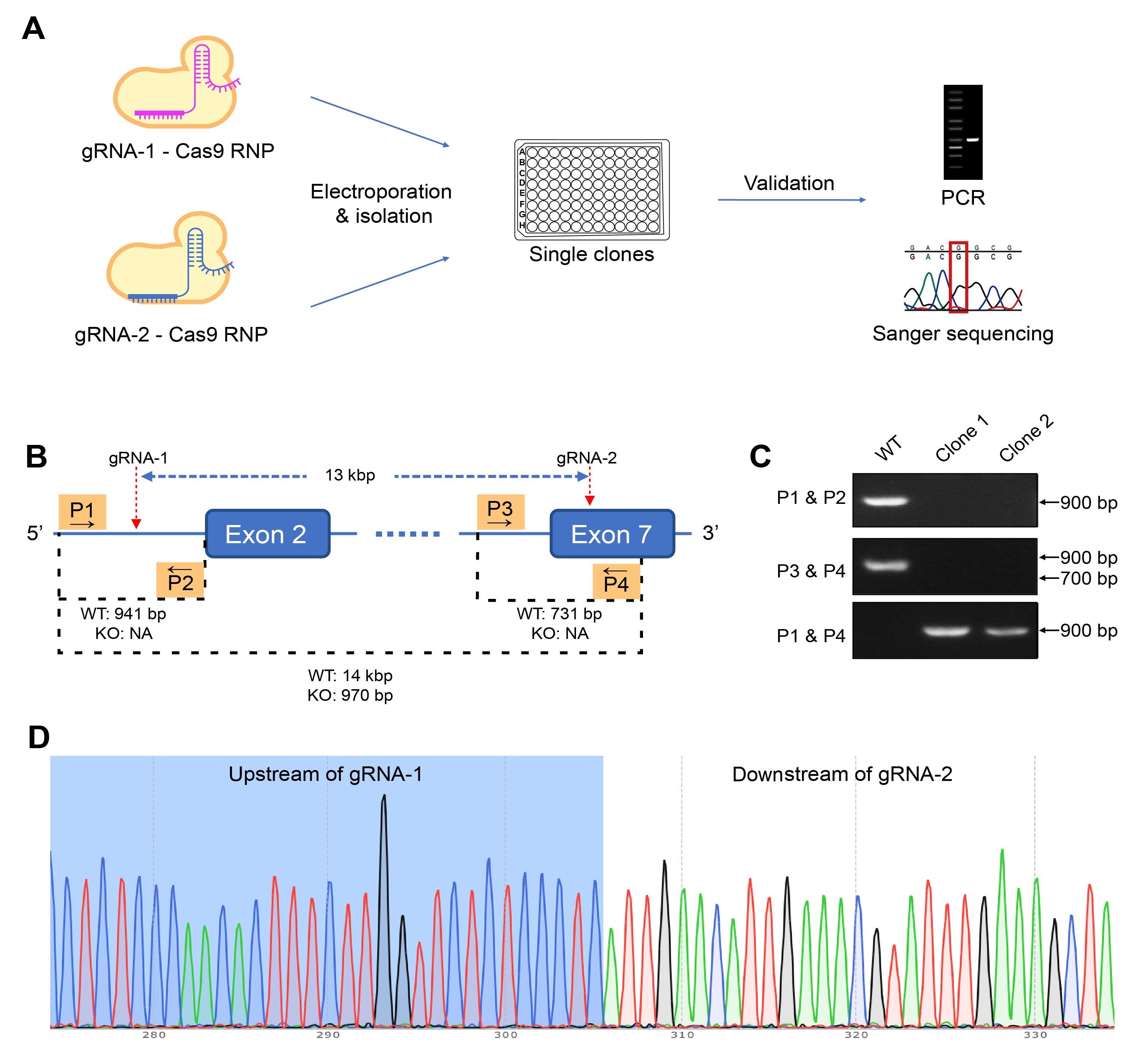
図3. gRNA-Cas9リボ核タンパク質(RNP)を用いたホモ接合体CD274ノックアウト(KO)変異体の作製。 (A)CRISPR編集用RNPを標的細胞にエレクトロポレーション、単一クローンを単離し、スクリーニングした。ノックアウト候補の遺伝子型はPCRとサンガーシーケンスを用いて検証した。(B)マウス結腸腺がん細胞株を編集した事例。標的遺伝子上の2つの部位をターゲットにするRNPを細胞内にエレクトロポレーションし、13kbpの領域をKOした。KOクローンとWTクローンを区別するために、P1からP4の4つのプライマーをデザインし、3回のPCRで検証した。(C)のPCRの結果から、クローン1はホモ接合性のKO体であることが確認され、さらに(D)のシークエンス結果からも確認された。
ご注文方法
リソース
FAQ
どちらの方法を採用するかは、標的細胞と遺伝子に大きく依します。シングルgRNAを用いたフレームシフト変異は、大抵の場合は遺伝子の機能破壊に十分です。同じ機能を持つ遺伝子がある場合は、配列欠失によって複数の遺伝子の断片をノックアウトする必要があります。
Cas9ニッケースが単一の標的遺伝子のそれぞれのDNA鎖を標的とする場合、デュアルgRNAを使用することができる。このアプローチでは、2つのgRNAが標的部位の両鎖のそれぞれに誘導され、ニッケースが一本鎖切断を生成した結果、標的部位にDSBが生じます。この方法は、DSBのために両方のgRNAによるターゲティングが必要であるため、CRISPR/Cas9発現のオフターゲット効果を低減する効果が期待できます。
目的遺伝子の働きを不活性化し、その影響を調べる目的ではshRNAノックダウンもCRISPRを使ったノックアウトのどちらも役に立ちます。目的に応じてどちらがより適しているかを判断するためのポイントを以下に記載します。
メカニズム
- ノックダウンベクター
ノックダウンベクターはshRNAという短いヘアピン状のRNAによって細胞の標的mRNAを分解、翻訳阻害することで目的遺伝子の機能を抑えます。そのため、shRNAノックダウンベクターは標的遺伝子配列を変えることはありません。
- ノックアウトベクター
CRISPRはヌクレアーゼにゲノム上の標的配列を切断するように働きかけます。分解された配列が不完全な形で修復され結果、配列の挿入もしくは欠損が起こり、恒久的な変異となります。それらの変異のいくつかは翻訳コドンのずれ(フレームシフト)やストップコドンを作り出し、標的遺伝子の機能不全を引き起こします。ゲノム上で数kb以内にふたつの標的配列が同時に切断された場合は、その間の配列に欠損が起こることもあります。
効率
shRNAノックダウンは最も効果的なshRNAをつかったとしても、標的遺伝子の発現を100%抑制することはできません。一方でCRISPRの場合、一部の細胞で遺伝子の機能を完全に欠損させる変異を持つ細胞を作成できます。
再現性と均一性
一般的にshRNAベクターは、処置を施した細胞プール内のほとんどの細胞から均一な結果が得られ、複数の実験の間で高い再現性が得られます。一方でCRISPRは変異が偶発的に引き起こされるので、得られる結果が細胞によって違います。細胞の標的遺伝子を完全に欠損させるためには細胞内のすべての遺伝子コピーを破壊しなくてはなりません。X-またはY-染色体上の遺伝子を除いて大抵の細胞は遺伝子が2コピーあり、そしてがん細胞は2コピー以上の遺伝子を持つので遺伝子を完全に欠損させた細胞が得られる可能性はとても低くなります。このような理由から、CRISPRのようなヌクレアーゼを利用した遺伝子ノックアウトはすべての標的遺伝子コピーが破壊された細胞をDNAシークエンスによって特定する必要があります。
オフターゲット効果
オフターゲット効果はshRNAノックダウンでもCRISPRノックアウトでも報告されています。観察された表現型がオフターゲット効果であるかどうかは同じ標的遺伝子に対して異なる複数のshRNAを使うことで確認できます。もし、異なる複数のshRNAノックダウンから得られた表現型が同じならば、その表現型はオフターゲット効果ではない証拠となります。CRISPRノックアウトを使う場合は、オフターゲット効果による表現型でないことを確認するために標的遺伝子に対して異なる機能欠損変異を持つ複数のクローンを作成して解析する必要があります。加えて、得られた変異体クローンのゲノムから、バイオインフォマティクスによって推測されるオフターゲットとなりうる配列をシークエンスして、それらに変異がないことを確認する方法もあります。




