shRNA ライブラリー(プール型)
VectorBuilderは、ヒトおよびマウスの遺伝子をターゲットとする高品質の既製品shRNAライブラリー(プール型)をご提供します。これらのRNAiライブラリーは、疾患経路、薬物治療に対する細胞応答、発生過程、遺伝子調節などに関与する遺伝子の大規模な機能欠損型スクリーニングを強力にかつ低価格に実行できるツールです。VectorBuilderは、さらにカスタムでご希望のターゲット遺伝子リストに基づいたカスタムshRNAライブラリーも受託構築いたします。
提供しているshRNAライブラリーの種類
- ヒト遺伝子をターゲットしたレンチウイルスライブラリー、既製品、プール型: エリート遺伝子群 (~2,000 種類のPubMed Centralで引用されているコモン遺伝子群)、ホールゲノム (>20,000 RefSeq genes)
- マウス遺伝子をターゲットしたレンチウイルスライブラリー、既製品、プール型: エリート遺伝子群 (~2,000 種類のPubMed Centralで引用されているコモン遺伝子群)、ホールゲノム (>20,000 RefSeq genes)
- カスタムshRNAライブラリー、プール型(納品形態:大腸菌グリセロールストック、プラスミドDNAプール、組み換えウイルスプール)
ご注文情報
価格と作業日数 プライスマッチ
| 製品名 | 遺伝子数 | shRNA数 | スケール | カタログ番号 | 価格 (消費税・送料別) | 作業日数 | |
|---|---|---|---|---|---|---|---|
| ヒト エリート遺伝子 プール型 shRNA ライブラリー | 2,161 | 12,471 |
中容量 (>1.0x108 TU/ml, 1 ml) |
LVM (Lib190505-1037bjk) | 310,000円 | 7-14 日 | |
| マウス エリート遺伝子 プール型 shRNA ライブラリー | 2,233 | 12,472 |
中容量 (>1.0x108 TU/ml, 1 ml) |
LVM (Lib190505-1039sgb) | 310,000円 | ||
| ヒト ホールゲノムプール型shRNA ライブラリー | 20,593 | 105,233 |
中容量 (>1.0x108 TU/ml, 1 ml) |
LVM (Lib230926-1079mym) | 310,000円 | ||
|
プラス (>1.0x108 TU/ml, 5 ml) |
LV5M (Lib230926-1079mym) | 387,500円 | |||||
| マウスホールゲノム プール型shRNA ライブラリー | 22,023 | 105,170 |
中容量 (>1.0x108 TU/ml, 1 ml) |
LVM (Lib230926-1080rpt) | 310,000円 | ||
|
プラス (>1.0x108 TU/ml, 5 ml) |
LV5M (Lib230926-1080rpt) | 387,500円 |
カスタムshRNAライブラリーをご検討の場合は、ここをクリックしてお問い合わせください。
価格は予告なく変更される場合があります。
デコンボリューションサービス
スクリーニングした細胞のshRNAヒットを同定するには、細胞からゲノミックDNAを抽出してVectorBuilderにサンプルを送付してください。VectorBuilderにてQCチェック、NGSライブラリー調整を行い、ハイスループットシークエンシングを行います。NGSサービスは、イルミナ NovaSeq プラットフォームを相乗り価格でご提供いたします。納品形態はクラウドダウンロードまたは、USB納品です。ダウンロードリンク先には、未処理NGSシークエンシングデータ、解析データが保存されています。解析終了後、数週間から1ヶ月、ユーザーデータをクラウドに無料で保存します。
デコンボリューションサービスのお見積り依頼は、VectorBuilderホームページの デザインサポートを依頼する からお受けしています。
技術的情報
製品の特長
NGSによるライブラリーの品質検証
ベクタービルダーのライブラリーは、次世代シーケンサー(NGS)による厳格な品質検証によって高いリードアライメント率と均一性の面で、卓越した性能を示しています。
全ゲノムをカバーし、高いターゲットカバー率を実現
ヒトとマウスともに、以下の2種類のスケールで shRNAライブラリーをご提供しています: ホールゲノム(~19,000 RefSeq genes) 、エリート遺伝子 (~2,000のPubMed Centralで高頻度で引用される遺伝子群)。ノックダウンを確実にし、そしてスクリーニング効率を上げるために、shRNAターゲットシークエンスは、高い ノックダウンスコアー を出すshRNAシークエンスをRNAi consortium (TRC)のガイドラインに沿って選出し、各遺伝子に対して平均して5-6種類の異なるターゲットshRNAをライブラリーに追加しています。ターゲット遺伝子リストと、各 shRNAs は “技術的資料” より公開しています。
高い均一性
プール型プラスミドライブラリーは次世代シークエンシング(NGS)によってshRNAターゲットの検証を実施しています。エリート遺伝子ライブラリーではデザインしたshRNAが100%検出されること、また、ホールゲノムライブラリーでは デザインしたshRNAが>97%以上の検出されること、さらにプラスミドライブラリーとして、shRNAの存在率が高い均一性を保持していることが弊社製品としての必須条件です (図 1)。
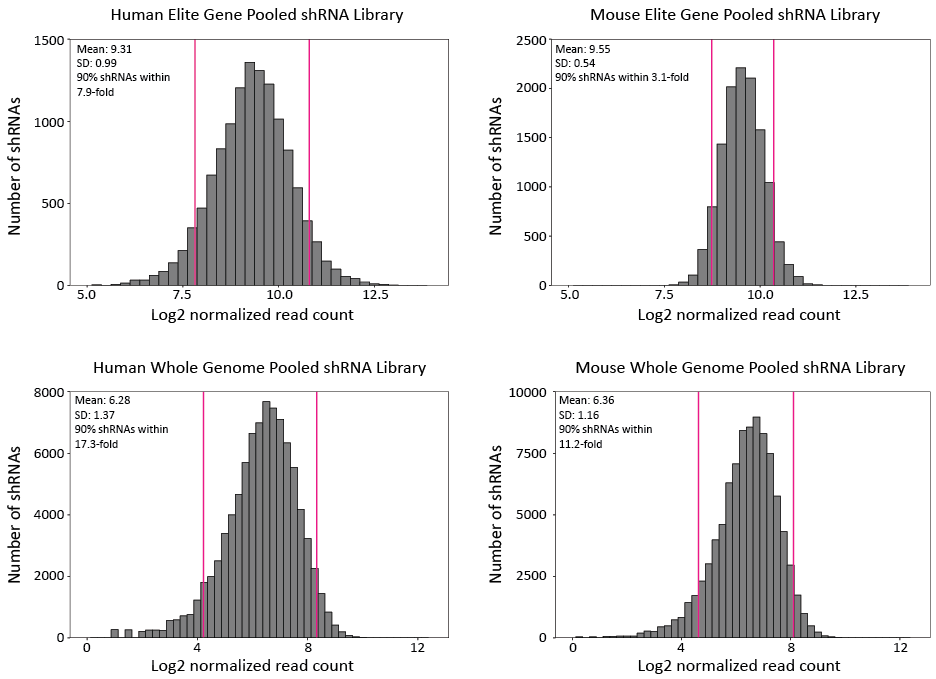
図 1. 異なるプール型ライブラリーのshRNAの存在率(representation)の比較。shRNA読み取り数は、NGSライブラリーサイズ(10 million reads)で平均化し、log2 scaleでプロットした。
すぐに実験に使えます!優れたレンチウイルスシステムをready-to-use高タイターで納品
プール型shRNAライブラリーは、第3世代のレンチウイルスベクターシステムをバックボーンにヒトU6プロモーターによって発現調節がされるようにデザインされています。ヒトU6プロモーターは様々な細胞株で、目的遺伝子のノックダウンを効率的に実施できるシステムです。レンチウイルスベクターシステムはshRNAを効率的に均一に、永久的に細胞に導入するため最適な in vitro遺伝学的スクリーニングを提供します。 全てのプール型 shRNAライブラリーは高い感染タイター (>108 TU/ml)を持ち、すぐに実験に使える(ready-to-use)レンチウイルスとして納品されるため、ウイルスパッケージングやタイター測定の手間がありません。さらに第3世代のレンチウイルスは、自己増殖能を欠損させているためバイオセイフティー上の安全性も向上しています。
図 2. 哺乳類用shRNAノックダウンレンチウイルスベクターのマップ
アノテーションされたshRNAライブラリーベクターのマップとシークエンスを見るレンチウイルス shRNAノックダウンベクターについてもっと読む
効果的で多目的な二重マーカーによる選択または形質導入された細胞の追跡
レンチウイルスベクターでは、二重マーカーとして EGFPとピューロマイシン(puromycin:Puro)耐性遺伝子が発現します。形質導入された細胞はピューロマイシン薬剤選択による生存とEGFP緑色蛍光の可視化によって追跡できます。

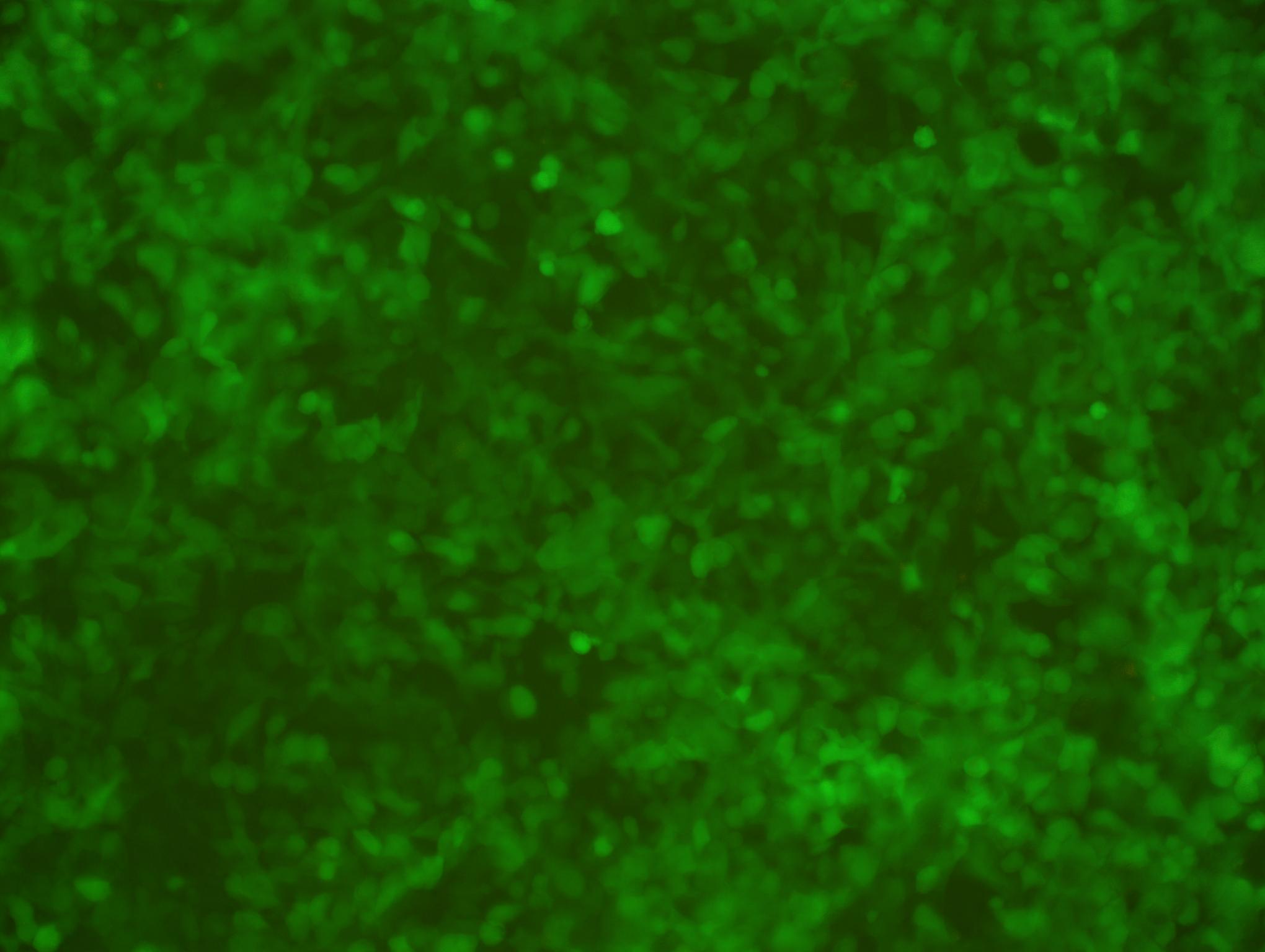
図 3. ヒトエリート遺伝子プール型shRNAライブラリー (MOI=10)の導入後、ピューロマイシン選択 (1.5 ug/ml)から4日後のEGFPを発現している293T細胞。倍率: 200x. 左: 明視野イメージ、右: 蛍光顕微鏡イメージ
プール型shRNAライブラリーを使用した遺伝学的スクリーニングのワークフロー
プール型shRNAレンチウイルスライブラリーを用いた標準的な遺伝学的スクリーニングのワークフローを 下の 図 4 に示します。まず第一に、shRNAレンチウイルスライブラリーを細胞に対して感染させます。レンチウイルスが感染した細胞を陽性マーカー(例:薬剤耐性遺伝子または蛍光タンパク質)によって選択します。次に、陽性細胞を、参考集団(reference population)と実験集団(experimental population)に分けます。次に実験集団に対して特定の選択圧を加え(例:薬剤処理や継代)興味ある表現型を持つ細胞を特定します。スクリーニング方法には次のメジャーな3種類があります: 1) 生存力スクリーニングといいshRNAがエンリッチメントまたは枯渇した細胞が、特定の選択圧に曝されても生存する細胞を探す方法; 2) レポーターの発現を指標にしてレポーター発現の高低をshRNAのエンリッチメントで探す方法(例:shRNAが転写因子をターゲットし、その結果としてレポーター遺伝子発現を変化させる場合など; 3)細胞のふるまいの変化をスクリーニングする方法で、shRNAが細胞浸潤、転移などにかかわる遺伝子をターゲットした場合の方法です。 スクリーニング後は、実験集団の細胞と、参考集団の細胞をそれぞれ回収し、サンガーシークエンシングまたは次世代シークエンシングによって、参考集団に対して、実験集団でエンリッチ、または枯渇された遺伝子を同定します。このようにスクリーニングされた候補遺伝子群は、さらに該当する機能解析を行い、解析を続けます。
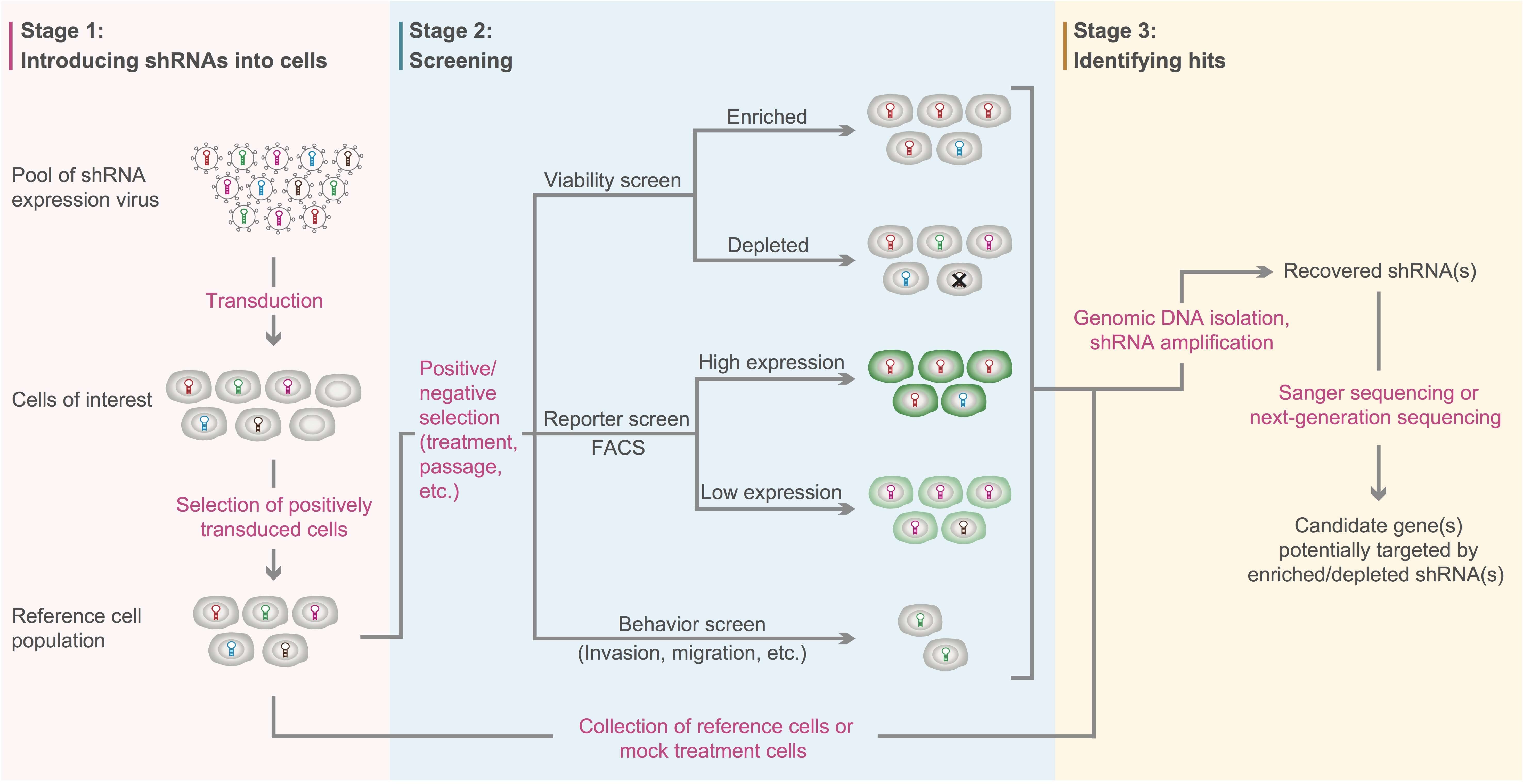
図 4. プール型 shRNA レンチウイルスライブラリーを使い、loss-of-functionスクリーニングを行う場合のワークフロー。Acta Biochim Biophys Sin 44:103-112 (2012)から引用
リソース
技術的資料
ターゲット遺伝子とshRNAの完全リスト
Q&A
プール型RNAiスクリーニングとアレイ型RNAiスクリーニングを比較した場合の利点は?
プール型RNAiスクリーニングは、アレイ型RNAiスクリーニングに対して数々の優れた点があります。下の表にまとめました:
| アレイ型スクリーニング | プール型RNAiスクリーニング | |
|---|---|---|
| shRNAはどのように目的細胞に導入されますか? | 個々のshRNAを、個々のマルチウエルプレートで培養された細胞にアプライする(例:96穴または384穴)。 | 数百~数千種の異なるshRNAを、細胞集団に同時にアプライする。 |
| shRNA効果による表現型はどのように識別しますか? | 興味ある表現型を示すウェルは、個々のウェルごとで表現型を調べ選択します。個々のウェルの shRNA の ID は既知です。 | 興味ある表現型を示す細胞を細胞集団から選択します。 shRNAがエンリッチまたは枯渇されたと想定される目的細胞は、シークエンスシングによって同定します。 |
| 遺伝子相互作用のスクリーニングは可能ですか? | 不可能 (単一のshRNAを各ウエルにアプライした場合)、または、限定的 (1種類以上のshRNAをユーザー側がミックスして各ウエルにアプライした場合)。 | 可能(細胞は複数のランダムな組み合わせのshRNAで遺伝子導入されることがあり、互いに遺伝学的相互作用のある遺伝子変異を持つ場合がある)。 |
| 技術的習熟度の違いによるデータのバリエーション | 高い | 低い |
| 試料のコストと労働費 | 高い | 低い |
| 特別な装置の必要性 | 高い(例:リキッドハンドラー、ハイスループット画像処理設備など) | 低い(ラボベンチ上の標準機器) |
shRNAノックダウンスコアーはどのように計算されていますか?
VectorBuilderでは、 RNAiコンソーシウム (TRC) が使用しているルールに似た方法に弊社独自の改変を加えてshRNAをデザインし、shRNAのノックダウンスコアーを出しています。同定されている RefSeq転写産物に対して、ターゲットサイトの候補となる全ての21merシークエンスを求めます。シークエンスは次の特長を持った場合、候補から除外されます: ノックダウンの効率/特異性を低下させると知られている特長を持ったシークエンス、クローニング作業が複雑度を増すシークエンス、さらに同一塩基が≥4連続、GまたはCが≥7連続する場合、GC 含量が25%以下または 60%以上の場合、さらに AAが5’ 末端にある場合などです。ノックダウンスコアー値は以下の様な場合、ペナルティーがついて値を下げていきます:内部にステムループ構造を持つ、3’末端に向かって高いGC含量を持つ、既知の miRNAシードシークエンスの場合、または他の遺伝子がオフターゲットとしてマッチする場合です。複数の転写産物が1遺伝子から合成される場合、全ての転写産物をターゲットするシークエンスは高いターゲットスコアーが与えられます。
全てのスコアーは ≥0以上で、中間値が ~5。標準偏差が~5。95%のスコアーは ≤15以下となります。ノックダウンスコアーが15に近いshRNAは、ベストなノックダウンパフォーマンスでクローニングも容易であると判断されます。対して、ノックダウンスコアーが0のshRNAの場合、ノックダウンパフォーマンスも貧弱で、クローニングも困難であると判断されます。
ただこのように求められら特異性スコアーはshRNAの特異性の大まかな指標として用いるようにしてください。実際の研究場面ではshRNAの特異性スコアーが低くても、実験では十分に使えるgRNAがある例もあり、また、ノックダウンスコアーが高いものの実際のパフォーマンスが貧弱な例も知られています。そして3’ UTR領域をターゲットとして選択することは、コーディンクシークエンスをターゲットするのと同様に効果的であることを念頭に置いてください。



