dual-gRNA CRISPRライブラリー(プール型)
VectorBuilderは高品質なCRISPRベースの、ヒトまたはマウスの全ゲノムノックアウトスクリーニング用に既製品 dual-gRNAレンチウイルスライブラリーをご提供しています。可能な限り、1ターゲット遺伝子に対して、リダンダントに 4-6種類の異なるgRNAペアーを、各ベクターにデザインしています。このライブラリーを使うことで、ゲノムワイドの機能欠損型スクリーニングを強力に、かつ省コストで実施できます。これら既製品ライブラリーに加え、VectorBuilderでは、ユーザーのニーズに合わせたカスタムgRNAライブラリーもも受託構築いたします。
ご提供しているdual-gRNA CRISPR ライブラリーの種類
- ヒトホールゲノム レンチウイルスライブラリー 既製品(Ready-to-use)、プール型 (20,048種 ヒト遺伝子)
- マウスホールゲノム レンチウイルスライブラリー 既製品(Ready-to-use)、プール型 (20,493種マウス遺伝子)
- カスタム gRNAライブラリー、プール型 (納品形態:大腸菌グリセロールストック、プラスミドDNA、または組み換えウイルス)
ご注文情報
価格と作業日数 プライスマッチ
| 製品名 | 遺伝子数 | gRNA ペアー数 | 容量 | カタログ番号 | 価格 (消費税、送料別) | 作業日数 | |
|---|---|---|---|---|---|---|---|
| ヒト ホールゲノム dual-gRNAライブラリー | 20,048 | 91,926 |
中容量 (>1.0x108 TU/ml, 1 ml) |
LVM(Lib190505-1046fgb) | 465,000円 | 7-14 日 | |
|
プラス (>1.0x108 TU/ml, 5 ml) |
LV5M(Lib190505-1046fgb) | 542,500円 | |||||
| マウス ホールゲノム dual-gRNAライブラリー | 20,493 | 90,344 |
中容量 (>1.0x108 TU/ml, 1 ml) |
LVM(Lib190505-1050kpm) | 465,000円 | ||
|
プラス (>1.0x108 TU/ml, 5 ml) |
LV5M(Lib190505-1050kpm) | 542,500円 |
カスタムライブラリーをご検討の場合は、ここをクリックして問い合わせてください。
価格は予告なく変更される場合があります。
デコンボリューションサービス
スクリーニングした細胞のgRNAヒットを同定するには、細胞からゲノミックDNAを抽出してVectorBuilderにお送りください。VectorBuilderの技術員が、NGSライブラリーを調整し、イルミナNovaSeqでハイスループットシークエンシングを行います。納品形態はクラウドダウンロードまたは、USB納品です。ダウンロードリンク先には、未処理NGSシークエンシングデータ、解析データが保存されています。解析終了後、数週間から1ヶ月、ユーザーデータをクラウドに無料で保存します。
デコンボリューションサービスのご依頼は、 デザインサポートを依頼する からお問合せください。
技術的情報
製品の特長
ユニークなdual-gRNAレンチウイルスベクターのデザイン
既製品として全ゲノムノックアウト用レンチウイルスdual-gRNAライブラリーを販売しているのは、世界的にもVectorBuilderだけです。レンチウイルスにはgRNAペアーの発現カセットとEGFP/puromycinの二重マーカーカセットが搭載されています (図 1)。同一ベクター上のペアーgRNAsは、細胞に導入されると、同時に発現が開始し、一つのターゲット遺伝子上の異なるサイトを認識し、2か所のCRISPR切断サイトを生じます。結果的に大きな欠失が形成され、ターゲット遺伝子の機能欠損型、ノックアウトスクリーニングが実施できます。2種類のgRNAは、それぞれヒトU6プロモーターまたはアカゲザル(macaque)U6プロモーター、そしてgRNAスキャフォールドシークエンス の下流に挿入されます。これらのコンポーネントの個々のシークエンスは異なりますが、機能的に相同です (参考:Nat Methods. 14:573 (2017)) 。そのため、2種のgRNAカセット間での不必要な相同組み換えを抑制しながら、上流と下流のカセット領域のPCRやNGSシークエンシングを行うことが出来、ライブラリー品質の検証が効果的に実施できるようにデザインされています。
図 1. dual-gRNAライブラリーのベクターマップ
アノテーションされたdual gRNAライブラリーベクターのマップとシークエンスを見る
全ゲノムをカバーし、高いターゲットカバー率を実現
既製品dual-gRNAレンチウイルスライブラリーは、ヒト遺伝子20,048種、マウス遺伝子20,493種をターゲット遺伝子として網羅しています。個々の遺伝子は、異なるgRNAのペアーから成る4-6種類のベクターでターゲットされています。gRNAは次のパラメーターで選択されています: 1) オフターゲット効果を最小にするために、オフターゲットスコアーを考慮t; 2) 遺伝子の機能欠損表現型を欠失によって確実に生じさせる切断サイトを選択; 3) 切断サイトの距離で変異にバイアスが生じないよう考慮。それぞれの切断サイトで局所的な変異が生じるよりも、大きな欠失が生じるようにデザインしています。そして 4) 複数の転写産物が1遺伝子座から転写されている場合は、ノックアウトする転写産物の数を最大にするように考慮。全てのgRNAが切断するサイトは同定されているエキソン内にデザインしています。そのため、万が一、大きな欠失を生じることができなかった場合でも、切断部位局所的な小さな欠失によって、遺伝子機能をできる限り壊せるようgRNAシークエンスを選択しています。ターゲット遺伝子とgRNAのリストは “技術的資料”からご覧いただけます。
ライブラリー品質はNGSにて検証済
全てのライブラリーは、ペアーとなっている2か所のgRNAシークエンスを読み、gRNAの均一性をNGSで検証しています。>85% (ヒト)または>75% (マウス)のトータルシークエンス結果は、デザインされた gRNA ペアーに一致しています。さらに、デザインされたgRNAの >99% (ヒト)または >97% (マウス) はNGSで同定されています。そのため、これらのライブラリーはデザインされたgRNAペアーを広くカバーし、エラーが低いことを示しています。 ベクタービルダーのdual-gRNAライブラリーは、今まで論文でレポ―トされた、どのdual-gRNAライブラリーに比べて、より高品質なものであると言えます。
高い均一性
下ヒストグラムに示すように、各ライブラリープールには、gRNAペアーが再現性良く均一に含まれています (図 2)。
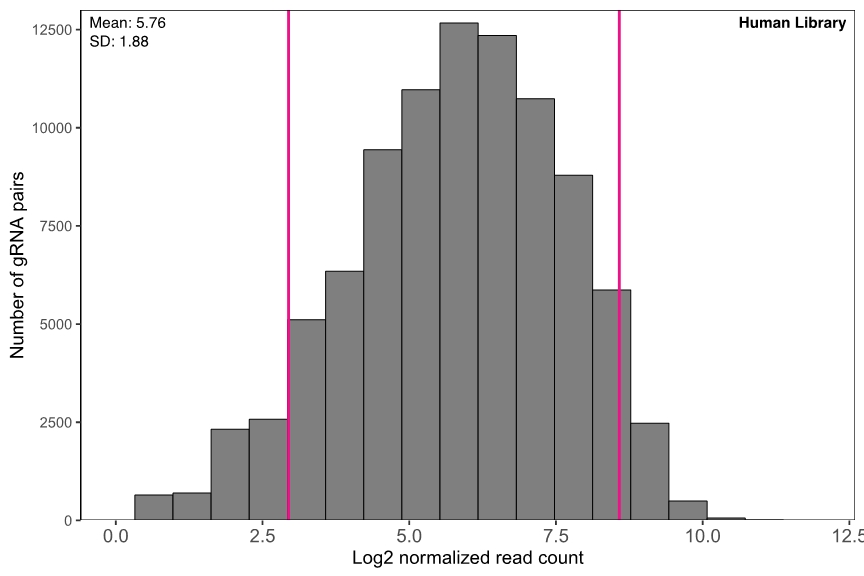
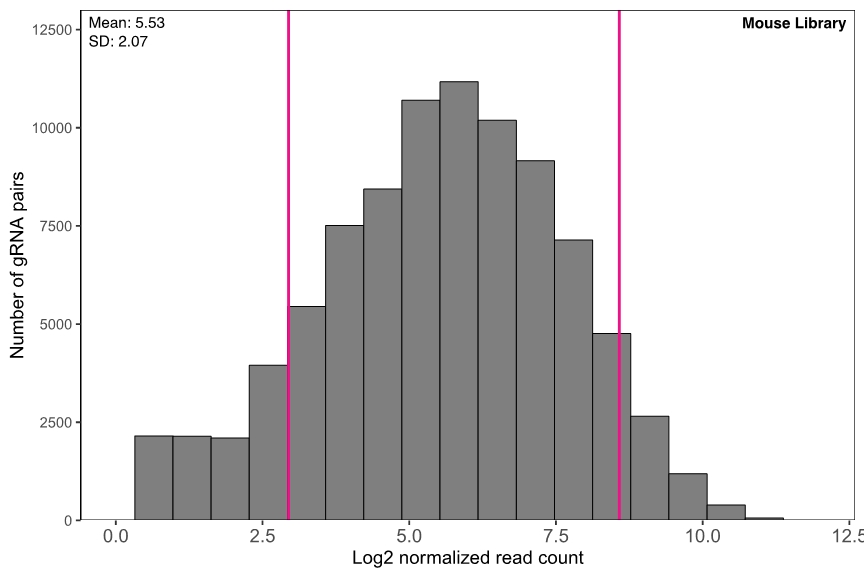
図 2. 異なるプール型ライブラリーのgRNAペアー存在率(representation)の比較 gRNA読み取り数はNGSライブラリーサイズ(10 million reads)で平均化し、log2 scaleでプロットした。
すぐに実験に使えます!優れたレンチウイルスシステムをready-to-use高タイターで納品
プール型CRISPRライブラリーは第3世代のレンチウイルスベクターシステムを使って確立されています。そのため、さまざまな細胞に対して高導入効率を発揮する発現システムとなっています。特に in vitro の遺伝学的スクリーニングには有効で、半永久的に効率よく、ほぼ均一にgRNAを細胞に導入します。全てのCRISPRライブラリーは、ready-to-use レンチウイルスで、機能的タイターが >108 TU/mlで納品されます。ウイルスパッケージングやタイター測定の作業なしに、すぐに目的のスクリーニングに使えることが魅力です。また、第3世代のレンチウイルスベクターシステムは、自己複製能力を欠損させているため、安全性も高められています。
形質導入された細胞を効率よく選択/追跡できる二重マーカー
レンチウイルスベクターバックボーンには、ピューロマイシン耐性遺伝子(Puro)とEGFPを発現するカセットが組み込まれています。そのため、導入細胞はピューロマイシンで薬剤選択し、緑色蛍光で可視化できます (図 3)。

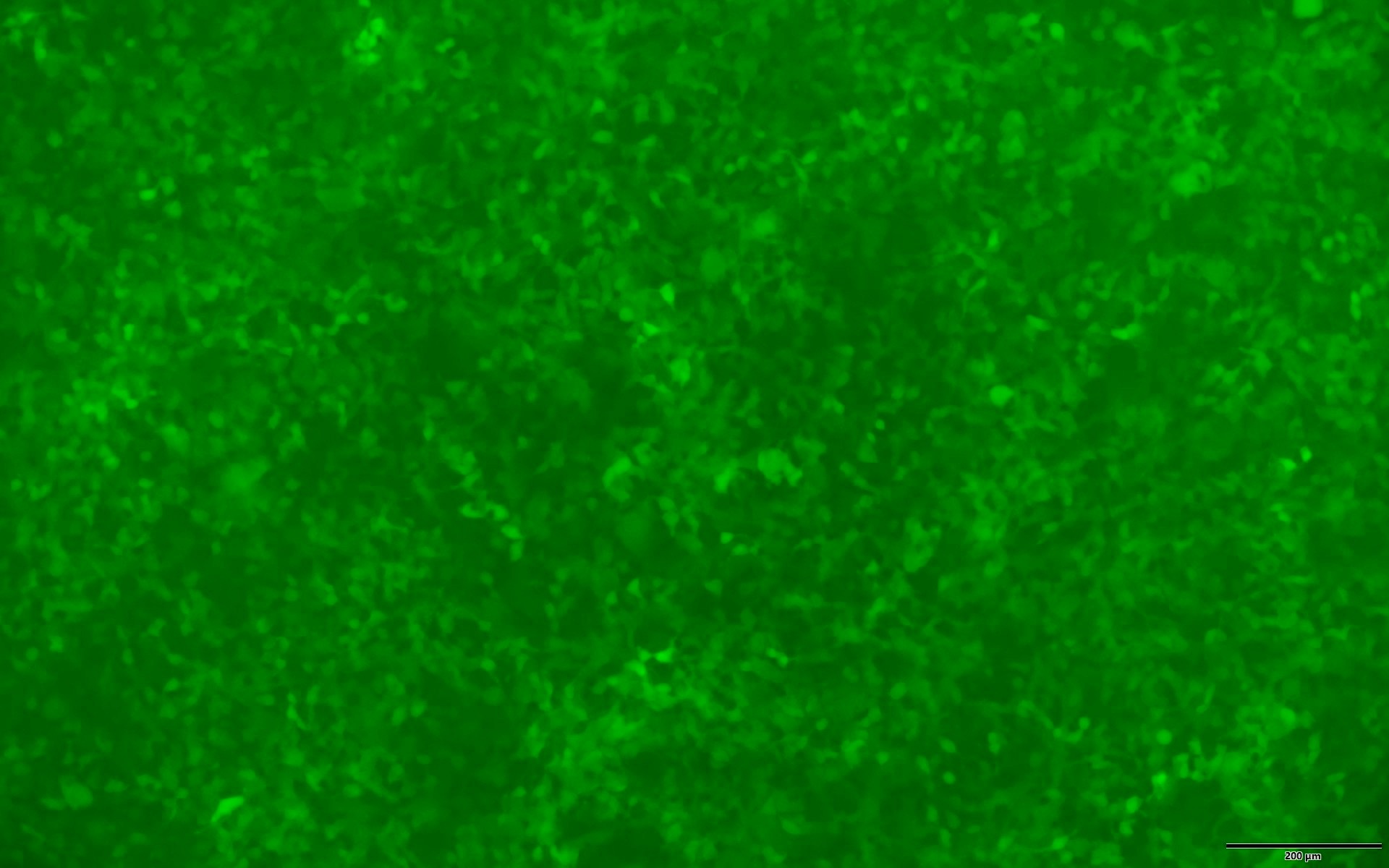
図 3. ヒト全ゲノム dual-gRNA レンチウイルスライブラリー(MOI=10)の導入後、ピューロマイシン選択 (1.5 ug/ml)4日後のEGFP を発現している293T細胞。 左: 明視野イメージ。 右: 蛍光顕微鏡イメージ
プール型CRISPRライブラリーを使用した遺伝学的スクリーニングのワークフロー
プール型デュアルgRNAレンチウイルスライブラリーを使った、遺伝学的スクリーニングワークフローの一例を 図 4に表します。まず、Cas9を発現する対象細胞にレンチウイルスライブラリーを感染させて目的の遺伝子導入を行います。レンチウイルスベクター上の選択マーカー(薬剤耐性や蛍光マーカーなど)で、陽性細胞を選択します。次に、陽性細胞をコントロール群と実験群の2つに分け、実験群にのみ特定の選択圧(例えば、薬剤処理や継代の繰り返しなど)を与えて、研究対象となる表現型を分離します。スクリーニングには大きく分けて3種類の方法があります:1) Viability 選択; 選択圧に晒された後、生存した細胞でgRNAのエンリッチメント(濃縮)またはデプリ―ション(枯渇)を検出する。2)レポーターによる選択; gRNAがエンリッチされた細胞でレポーター遺伝子の発現レベルが上昇、または低下することを指標としたスクリーニング(例えば、gRNAが転写因子をターゲットし、マーカー発現を変化させる場合など)。 3) behavior 選択; gRNAが細胞の浸潤、マイグレーションなどの運動性に関与している場合の選択。スクリーニングの後、実験群とコントロール群の両細胞群をハーベストし、実験群でエンリッチまたはデプリートされたgRNAをサンガーシークエンスまたはNGSでコントロール群と比較します。候補遺伝子を絞り込み、さらに関連する機能解析実験によって候補遺伝子の特定を進めます。
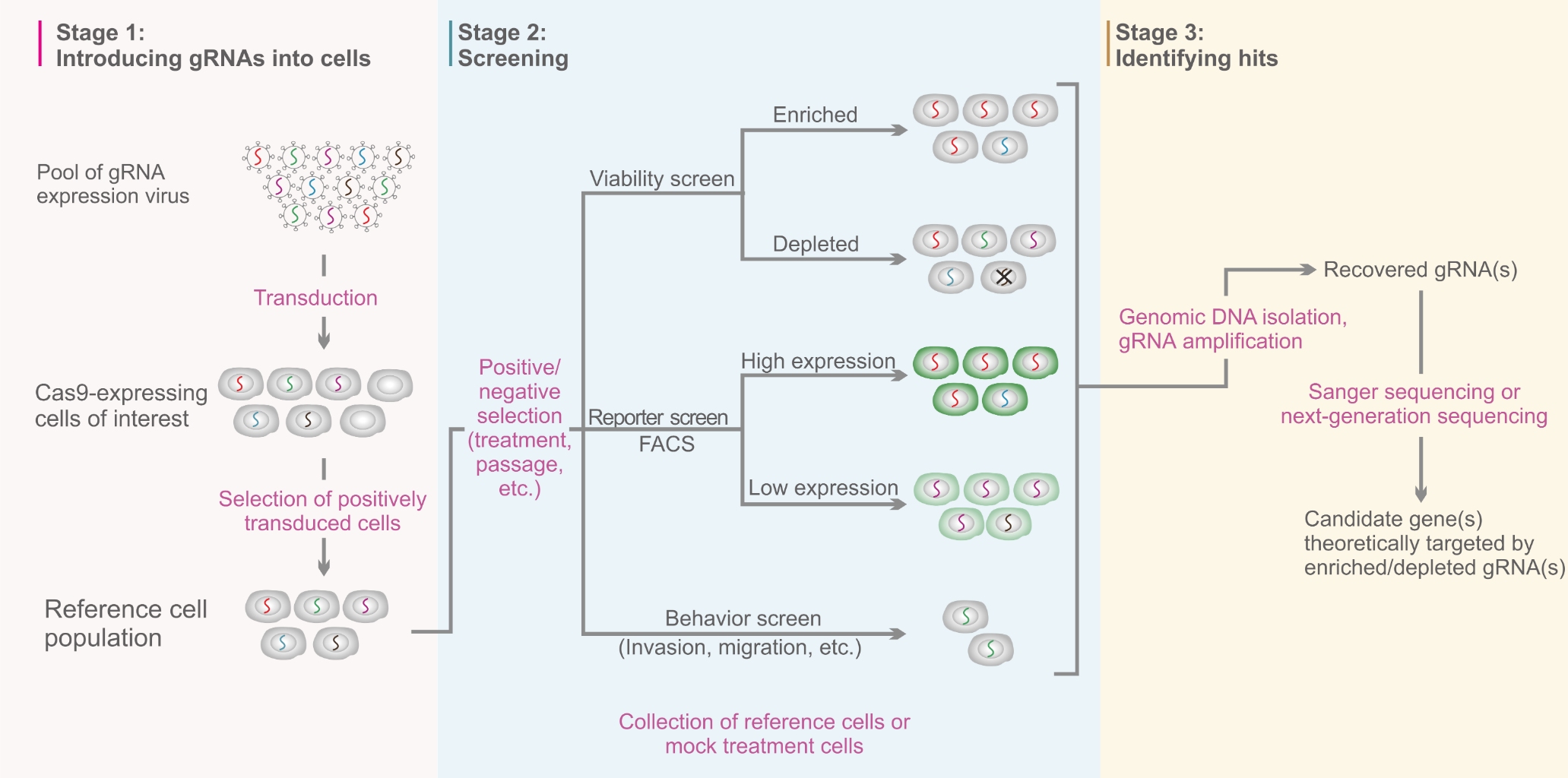
図 4. プール型dual-RNAレンチウイルスライブラリーを使ったCRISPRノックアウトスクリーニングのワークフロー。 Acta Biochim Biophys Sin 44:103-112 (2012)より引用。
技術的資料
ターゲット遺伝子とgRNAペアーの完全リスト
カタログとフライヤー
Q&A
シングル-gRNAライブラリーではなくデュアル-gRNAライブラリーを使う利点を教えてください?
Dual-gRNAライブラリーは、ノックアウトスクリーニングの実行に関してsingle-gRNAライブラリーが比較にならないほど強力です。なぜならdual-gRNAが生じさせる欠失は、ターゲット遺伝子の機能欠損変異を高頻度で生じさせるからです。Dual-gRNAライブラリーにプールされている各CRISPRベクターは、同一ターゲット遺伝子に対して一対(ペアー)の gRNAがクローニングされています。 Cas9発現細胞に、レンチウイルスによってこれらのCRISPRベクターが導入された場合、各ベクターは、同一遺伝子内の2か所のターゲットサイトを認識し、それぞれでDNAの切断を生じさせます。DNA切断が起きた細胞は、ダメージを修復しようとして切断サイト同士を繋げますが、この際に遺伝子内に大きな欠失が生じます。VectorBuilderでは、2か所のgRNAは、ターゲット遺伝子の機能的に重要な領域にわたるようにデザインされているため、結果的にDNAの切断・修復によって生じた欠失が、遺伝子機能欠損を招きます。
CRISPRノックアウトスクリーニングは、 1ベクターまたは2ベクターシステムのどちらが適しますか?
プール型CRISPRライブラリーの一般的なノックアウトスクリーニングでは、1ベクターシステム、または2ベクターシステムのどちらかのシステムフォーマットを使うように構築されます。1ベクターシステムでは、Cas9またはCas9変異型をgRNAと同一ベクター内にデザインしてベクターが構築されます。対して、2ベクターシステムでは、Cas9またはCas9変異型発現ベクターとgRNA発現ベクターの2種類のベクターが独立に構築されます。2ベクターシステムの変法としては、gRNA発現ベクターを、あらかじめ樹立したCas9安定発現細胞株に導入する方法もあります。1ベクターシステムの優れた点は、2ベクターの共トランスフェクションや共遺伝子導入を避けること、そしてCas9発現細胞株の樹立が不必要であることです。弱点としては2ベクターシステムに比べて、融通性に劣り、ウイルスのパッケージングサイズ限界のためにクローニングできる遺伝子サイズに制限がでてしまうことです。
gRNAの特異性スコアーはどのように計算されていますか?
VectorBuilderはgRNA特異性スコアの計算にCRISPRライブラリーデザイン(CLD)のアルゴリズムを使用しています。 このアルゴリズムでは、ある生物種のゲノムで、N(20)NGGシークエンスをターゲットとするgRNAがあるとすると、その生物種のゲノム中に、ターゲットシークエンスに対して≤3 のミスマッチを許した全てのオフターゲット候補を抽出します。そして各抽出オフターゲットに対してオフターゲットスコアーを計算します。gRNAの特異性スコアーの計算には、こうして抽出された全てのオフターゲットスコアーのアグリゲートを用います。gRNA特異性スコアーは、0から100の値で表示され、値が大きいほどターゲット特異性が高いことを示しています。
ただこのように求められら特異性スコアーはgRNAの特異性の大まかな指標として用いるようにしてください。実際の研究場面ではgRNAの特異性スコアーが低くても、実験上、十分に使えるgRNAがある例もあります。



